serverName <- "https://mocoriba.fr/mocoribaAPI/"Documentation de l’API MoCoRiBA

Cette documentation décrit le fonctionnement technique de l’API MoCoRiBA, qui alimente l’ensemble des visualisations présentes dans l’outil Viz.
Cette documentation vient en complément de la documentation “Données et méthodes - Projet MoCoRiBA-GC”. Celle-ci présente le projet et détaille les données utilisées, les principes appliqués ainsi que les modèles employés pour obtenir les résultats. Ces éléments ne seront pas abordés dans la présente documentation.
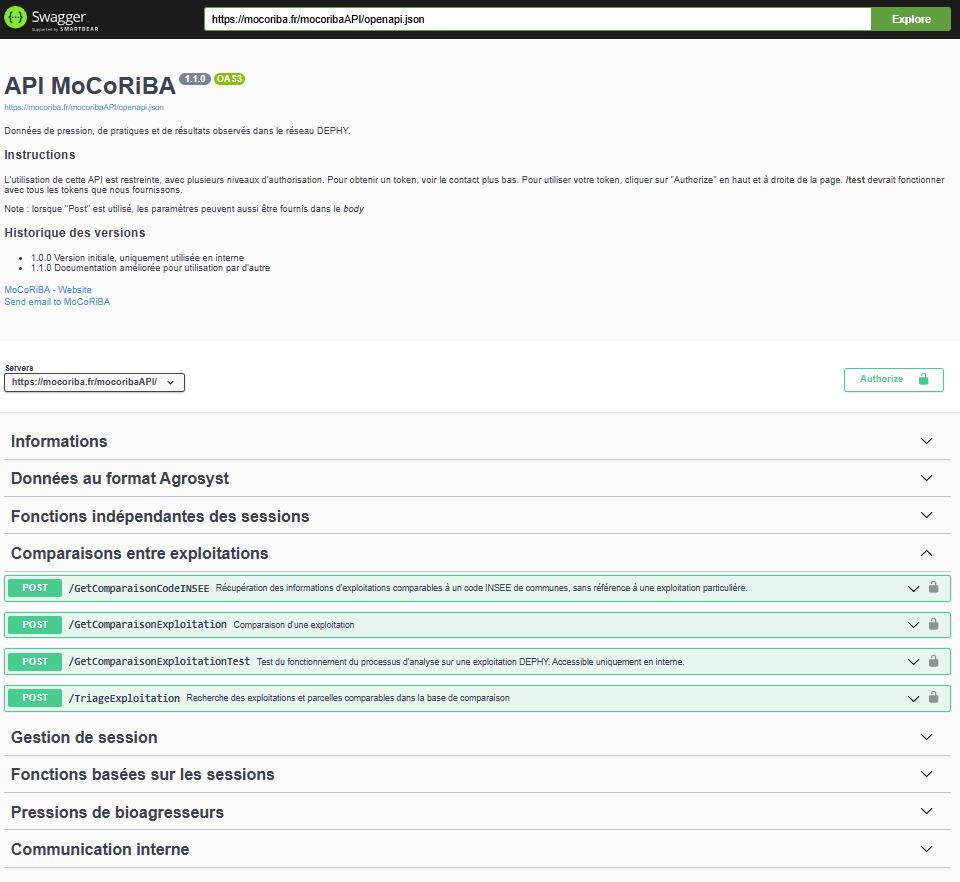
1 Swagger de l’API MoCoRiBA
Adresse du swager : https://mocoriba.fr/mocoribaAPI/__docs__/
Le Swagger de l’API MoCoRiBA, outil d’OpenAPI, est une documentation interactive qui décrit en détail l’ensemble des fonctionnalités disponibles dans l’API. Cette interface web permet de comprendre, tester et interagir avec l’API sans avoir à écrire de code manuellement. Elle répertorie toutes les fonctions (ou endpoints), en précisant pour chacune son nom, son rôle et ses paramètres d’entrée.
Des valeurs par défaut sont pré-remplies pour faciliter les tests, et il est possible de les modifier afin de simuler différents scénarios. Après l’exécution d’une requête, Swagger affiche la réponse de l’API, ce qui permet de visualiser les données retournées et d’en analyser le format.
Notre API est sécurisé par une authentification en deux étapes :
- Basic-Auth pour laquelle il faut entrer un login et un mot de passe.
- Un token qui permet d’utiliser l’API et qui est utilisé par l’API pour permettre l’accès à certaines fonctions et pas à d’autres.
Les deux authentifications sont nécessaires pour pouvoir utiliser l’API.
N’hésitez pas à nous demander un identifiant et un mot de passe pour accéder à l’API, en précisant vos besoins !
Pour vous connecter au Swagger de l’API MoCoRiBA vous devez d’abord entrer les login et mot de passe qui vous ont été fournis.
Ensuite, il faut déverouiller l’utilisation des fonctions avec l’aide du bouton “Authorize” qui ouvre la fenêtre suivante :

Vous pouvez ensuite essayer les fonctions, sans oublier de cliquer sur le bouton “Try it Out” avant d’entrer des paramètres :

2 Accèder à l’API MoCoRiBA via une interface R
L’objectif est de fournir les informations nécessaires pour utiliser l’API MoCoRiBA via une interface en langage R.
2.1 Paramètres de connexion à l’API
Adresse du serveur de l’API MoCoRiBA :
Utiliser les identifiants et password donnés pour Basic auth et le token vous autorisant l’accès à certaines fonctionnalité de l’API.
identifiant <- "**********"
password <- "**********"
tokenFunctionsAPI <- "**********"2.2 Fonctions génériques pour interroger l’API MoCoRiBA
L’API MoCoRiBA propose deux types de fonctions :
- Les fonctions de type ‘GET’.
- Les fonctions de type ‘POST’.
Pour simplifier les appels à l’ensemble des fonctions de l’API, nous mettons à votre disposition deux fonctions génériques. Celles-ci vous permettent, pour chaque appel, de spécifier uniquement le nom de la fonction et ses paramètres.
Pour cela vous avez besoin des librairies suivantes :
if(!requireNamespace("httr", quietly = TRUE)) {
install.packages("httr")
}
if(!requireNamespace("jsonlite", quietly = TRUE)) {
install.packages("jsonlite")
}
library(httr)
library(jsonlite)Pour les fonctions de type GET voici la fonction générique BasicGetMoCoRiBA :
Il vous suffira de fournir le nom de la fonction ainsi que les paramètres nécessaires.
Attention : Les paramètres qui ne contiennent pas une valeur unique (comme les listes ou les tableaux) doivent être convertis au format JSON.
BasicGetMoCoRiBA <- function(functionName, # nom de la fonction dans l'API
... # arguments avec leur nom et au format Json pour les listes d'éléments
){
Url <- paste0(serverName,functionName)
my_raw_result <- httr::GET(Url,
httr::authenticate(identifiant,password),
query=list(...))
basic_content <- httr::content(my_raw_result, as = 'text',encoding="UTF-8")
my_content_from_json <- try(jsonlite::fromJSON(basic_content),silent=TRUE)
if(class(my_content_from_json) == "try-error"){
out <- basic_content
class(out) <- "try-error"
}else{
out <- my_content_from_json
if(!is.atomic(out) && !is.null(out$error)){ # test if error only if not atomic
class(out) <- "try-error"
}
}
return(out)
}Pour les fonctions de type POST :
Il vous suffira de fournir le nom de la fonction ainsi que ses paramètres sous forme de liste dans le corps (body) de la requête.
Attention : Le token qui vous autorise à utiliser la fonction ne doit pas être inclus dans le body. Il doit être transmis séparément.
BasicPostMoCoRiBA <- function(functionName, # nom de la fonction dans l'API
body, # liste des arguments à mettre dans le body
...){ # arguments avec leur nom
Url <- paste0(serverName,functionName)
my_raw_result <- httr::POST(Url,
httr::authenticate(identifiant,password),
query=list(...),
body=body,
encode="json")
basic_content <- httr::content(my_raw_result, as = 'text',encoding="UTF-8")
my_content_from_json <- try(jsonlite::fromJSON(basic_content),silent=TRUE)
if(class(my_content_from_json) == "try-error"){
out <- basic_content
class(out) <- "try-error"
}else{
out <- my_content_from_json
if(!is.atomic(out) && !is.null(out$error)){ # test if error only if not atomic
class(out) <- "try-error"
}
}
return(out)
}Exemple d’utilisation de la fonction BasicGetMoCoRiBA
BasicGetMoCoRiBA("test",
entree="Test de l'API MoCoRiBA",
token=tokenFunctionsAPI)$msg
[1] "Bienvenue sur l'API MoCoRiBA, vous avez entré:'Test de l'API MoCoRiBA'"
$class
[1] "character"3 Se comparer aux fermes du réseau de DEPHY
L’objectif est de vous montrer comment enchaîner les différentes fonctionnalités de l’API, à travers un exemple concret. Celui-ci illustre une comparaison au niveau d’une exploitation, pour deux cultures données et deux campagnes spécifiques.
Pour récupérer les informations sur les performances des exploitations DEPHY comparables dans un contexte donné, il est nécessaire de disposer d’un dataframe contenant au minimum les informations de base sur l’exploitation de référence, au format Agrosyst.
Deux options sont possibles pour construire ce dataframe :
- À partir des données complètes de l’exploitation de référence (format AgroSyst) : Cela permet de comparer les performances de cette exploitation avec celles de ses exploitations similaires.
- À partir d’une exploitation dite “fictive”, ne contenant que : la position géographique (commune de l’exploitation), les cultures et les années concernées. Cette approche permet de récupérer uniquement les informations sur les exploitations comparables, sans référence à une exploitation réelle.
De plus, la comparaison à certaines échelle peut-être restreinte à certaines cultures et/ou campagnes de culture.
3.1 Fonctions d’informations sur les possibilitées de comparaison
L’API ne permet pas de comparer individuellement les performances de l’ensemble des cultures et destinations. Pour connaître la liste des cultures et destinations possibles, utilisez la fonction /culturesDisponible.
À noter : Une analyse au niveau des systèmes de culture prend en compte les performances de toutes les cultures possibles.
BasicGetMoCoRiBA("culturesDisponibles",
token = tokenFunctionsAPI)$culturesDisponibles
[1] "Avoine d'hiver (Grain)" "Avoine de printemps (Grain)"
[3] "Betterave (Sucre)" "Blé dur d'hiver (Grain)"
[5] "Blé tendre d'hiver (Grain)" "Blé tendre de printemps (Grain)"
[7] "Colza d'hiver (Grain)" "Lentille (Grain)"
[9] "Lin (Fibre)" "Lin (Grain)"
[11] "Luzerne (Ensilage)" "Luzerne (Fourrage)"
[13] "Maïs (Ensilage)" "Maïs (Grain)"
[15] "Orge d'hiver (Grain)" "Orge de printemps (Grain)"
[17] "Pois d'hiver (Grain)" "Pois de printemps (Grain)"
[19] "Pomme de terre (Tubercule)" "Seigle d'hiver (Grain)"
[21] "Soja (Grain)" "Sorgho (Grain)"
[23] "Tournesol (Grain)" "Triticale (Grain)" De la même manière, toutes les campagnes de culture ne sont pas disponibles. Pour chaque culture et pour une comparaison au niveau du système de culture, la fonction /culturesDisponiblesAnnees détaille les campagnes disponibles par culture.
À noter : Les dernières campagnes de culture sont ajoutées progressivement, au fur et à mesure de la réception des données Agrosyst et météorologiques.
cultCampDispo <- BasicGetMoCoRiBA("culturesDisponiblesAnnees",
token = tokenFunctionsAPI)
print(cultCampDispo[c(1:4,length(cultCampDispo))])$culturesDisponiblesAnnees
$culturesDisponiblesAnnees$`Avoine d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Avoine de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Betterave (Sucre)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Blé dur d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Blé tendre d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Blé tendre de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Chanvre (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Colza d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Lentille (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Lin (Fibre)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Lin (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
$culturesDisponiblesAnnees$`Luzerne (Ensilage)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Luzerne (Fourrage)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Maïs (Ensilage)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Maïs (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Orge d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Orge de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Pois d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Pois de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Pomme de terre (Tubercule)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Seigle d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Soja (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Sorgho (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Tournesol (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Triticale (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Système de culture`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$<NA>
NULL
$<NA>
NULL
$<NA>
NULL
$culturesDisponiblesAnnees
$culturesDisponiblesAnnees$`Avoine d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Avoine de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Betterave (Sucre)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Blé dur d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Blé tendre d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Blé tendre de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Chanvre (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Colza d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Lentille (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Lin (Fibre)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Lin (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
$culturesDisponiblesAnnees$`Luzerne (Ensilage)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Luzerne (Fourrage)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Maïs (Ensilage)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Maïs (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Orge d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Orge de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Pois d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Pois de printemps (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Pomme de terre (Tubercule)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Seigle d'hiver (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Soja (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Sorgho (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Tournesol (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Triticale (Grain)`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 2025
$culturesDisponiblesAnnees$`Système de culture`
[1] 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020 2021 2022 2023 2024
[16] 20253.2 Définition de la référence (réelle ou ‘fictive’)
Deux méthode sont possible pour se comparer à DEPHY, soit vous souhaiter mettre directement les données des pratiques de l’exploitation de référence au regard des résultats dans DEPHY, soit seul les résultats dans DEPHY vous intéresse pour une situation géographique ou pédo-climatique donnée. Pour cela, vous pouvez soit récupérer l’exploitation de référence directement via notre API si vous êtes partenaires ou reconstituer l’exploitation de référence à partir de vos données et sur une base Agrosyst, soit utiliser une exploitation dite ‘fictive’.
Il existe deux méthodes pour comparer vos données à celles de DEPHY :
- Comparaison directe : Vous souhaitez mettre en regard les pratiques de votre exploitation de référence avec les résultats disponibles dans DEPHY.
- Comparaison par contexte géographique ou pédo-climatique : Seuls les résultats DEPHY vous intéressent pour une situation géographique ou pédo-climatique donnée.
Pour cela, vous avez trois options :
- Récupérer l’exploitation de référence directement via notre API (si vous êtes partenaire).
- Utiliser une exploitation dite “fictive”.
- Reconstituer l’exploitation de référence à partir de vos propres données, en vous basant sur une exploitation dite ‘fictive’.
3.2.1 Données réelles de l’exploitation via l’API MoCoRiBA
L’utilisation d’une exploitation réelle nécessite de disposer des données au format Agrosyst. Pour cela, nous mettons à disposition des fonctions à usage restreint permettant de récupérer directement les données d’une exploitation au bon format :
- La fonction /GetAgrosystTableForWiuzIdExp : Elle permet de télécharger les données d’une exploitation de la coopérative Euralis au format Agrosyst, directement depuis l’API de WIUZ, à partir de son identifiant.
- La fonction /GetAgrosystFarmData : Elle permet de télécharger les données d’une exploitation du réseau DEPHY (base de données Agrosyst) au format Agrosyst, directement depuis nos serveurs.
Pour les exploitations Euralis :
# Identifiant Wiuz de l'exploitation Euralis
exploitation_id <- "**********"
datExp <- BasicGetMoCoRiBA("GetAgrosystTableForWiuzIdExp",
idExp = exploitation_id,
token = tokenFunctionsAPI)Pour les exploitations du réseau DEPHY :
# Identifiant Agrosyst de l'exploitation DEPHY
exploitation_id <- "**********"
datExp <- BasicGetMoCoRiBA("GetAgrosystFarmData",
idExp = exploitation_id,
token = tokenFunctionsAPI)Ces fonctions retournent 3 tables :{#descriptionTableExpRef}
- tableAnnuelle : synthèse des pratiques réalisées sur chaque parcelle par campagne.
- tableDetailIntrants : données de traitements phyto-pharmaceutiques par mois de l’année.
- tableDetailPhytoSubstancesActives : données de traitements phyto-pharmaceutiques à la substance active par mois de l’année.
List of 3
$ tableAnnuelle :Classes ‘data.table’ and 'data.frame'
..$ domaine_code : chr "**********", # Id de l'exploitation
..$ domaine_nom : chr "**********", # Nom de l'exploitation
..$ sdc_code : chr "**********", # Id du système de culture
..$ zone_id : chr "**********", # Id de la parcelle
..$ long : num "*****" # Longitude de la parcelles
..$ lat : num "*****" # Latitude de la parcelles
..$ domaine_campagne : int 2020, # Année de récolte (campagne)
..$ grande.région : chr "sud-ouest", # Région agro-climatique, voir (https://mocoriba.fr/documentation/donnees_methodes_MoCoRiBAviz.html#GR_agroCLim)
..$ codeGrandeRegion : num 11, # Id région agro-climatique
..$ codeDepartement : chr "64", # Code du département
..$ INSEE_COM : chr "64554", # Code INSEE de la commune
..$ zone_surface : num 5.01, # Surface de la parcelle (en hectares)
..$ culture_agregee : chr "Maïs", # Culture d'après le référentiel Agrosyst
..$ destination_simple_cor : chr "Grain", # Destination de la récolte d'après le référentiel Agrosyst
..$ culture_destination : chr "Maïs (Grain)", # culture_agregee (destination_simple_cor),
..$ culture_type : chr "PRINCIPALE" # Type culture (principale, intermédiaire, IAE, jachère,...)
..$ precedent_agregee : chr "Féverole d'hiver", # Culture précédente au format Agrosyst
..$ N_miner_Year : num 0, # Apports d'azote minérale en unités d'azote
..$ N_org_Year : num 50, # Apports d'azote organique en unités d'azote
..$ FERT_N_total : num 50, # Apports d'azote organique + minérale
..$ TILL_Labour : num 1, # Nombre de labours
..$ TILL_nbAvSemis : num 1, # Nombre de passages de travail du sol avant semis (sauf labour)
..$ TILL_nbApSemis : num 0, # Nombre de passages de travail du sol après semis
..$ TILL_total : num 2, # Nombre total de travail du sol
..$ irrigation : chr "non", # Culture irriguée ou non
..$ sum_irrigation_zone_cor : num NA, # Quantité d'eau apporté (en mm)
..$ rendZone : num 110, # Rendement de la culture
..$ unite_cor : chr "q/ha", # Unité du rendement de la culture
..$ date_recolte : Date "2020-10-30", # Date de récolte
..$ indice_Shannon_sdcCamp : num 0.683, # Diversité cultivée dans le système de culture pour cette campagne (indice de Shannon)
..$ exp_indice_Shannon_sdcCamp: num 1.98, # Exponnentiel de l'indice de Shannon (homogène à un nombre de culture présente à proportion également dans le système)
..$ IFT_INS_Year_Agrosyst : num 1, # IFT insecticides annuel (recalculé pour être homogène à Agrosyst)
..$ IFT_HER_Year_Agrosyst : num 1.35, # IFT herbicides annuel (recalculé pour être homogène à Agrosyst)
..$ IFT_FUN_Year_Agrosyst : num 0, # IFT fongicides annuel (recalculé pour être homogène à Agrosyst)
..$ IFT_n_pass_total : num 2, # Nombres de passages herbicides, fongicides ou insecticides
..$ IFT_total : num 2.35, # IFT insecticides + fongicides + herbicides
$ tableDetailIntrants :'data.frame':
..$ zone_id : chr "**********", # Id de la parcelle
..$ zone_surface: num 0.96, # Surface de la parcelle (en hectares)
..$ mois : int 5, # Mois (janvier=1, ...)
..$ campagne : int 2023, # Année de récolte (campagne)
..$ INSECTICIDAL: num 0, # IFT insecticides
..$ HERBICIDAL : num 0, # IFT herbicides
..$ FUNGICIDAL : num 0, # IFT fongicides
$ tableDetailPhytoSubstancesActives:'data.frame':
..$ zone_id : chr "**********", # Id de la parcelle
..$ mois : int 4, # Mois (janvier=1, ...)
..$ campagne : int 2020, # Année de récolte (campagne)
..$ intrant_phyto_type: chr "HERBICIDAL", # Type de produit phyto-pharmaceutique
..$ nom_sa : chr "benoxacor", # Nom de la substance active utilisée
..$ dose_sa_G_HA : num 90, # Quantité épandue par hectare (g/ha)3.2.2 Exploitation ‘fictive’ et structure de la base Agrosyst
Il est possible d’utiliser une exploitation dite ‘fictive’, identifiée uniquement par son code INSEE de commune et sans donnée de pratique, à l’aide de la fonction /basicAgrosystLine.
INSEE_COM <- "78615"
campagnes <- c(2021:2022)
culture_destination <- c("Blé tendre d'hiver (Grain)","Tournesol (Grain)")
datExpFictive <- BasicGetMoCoRiBA("basicAgrosystLine",
code_commune = INSEE_COM,
campagne = jsonlite::toJSON(campagnes),
agrosystCrop = jsonlite::toJSON(culture_destination),
token = tokenFunctionsAPI)
str(datExpFictive,1)List of 1
$ basicAgrosystLine:'data.frame': 4 obs. of 15 variables:Cette table de données contient le strict minimum pour être directement utilisée par les fonctions de comparaison de la partie suivante. Elle peut également servir de base pour reconstituer la table d’une véritable exploitation, en y ajoutant des informations sur les pratiques culturales.
À noter: La structure du format Agrosyst est disponible au travers de la fonction /StructureBaseAgrosyst. Elle décrit l’ensemble des variables pouvant être documentées ainsi que les valeurs possibles pour les variables qualitatives (liste non exhaustive pour certaines variables).
structure <- BasicGetMoCoRiBA("StructureBaseAgrosyst",
token = tokenFunctionsAPI)
str(structure,3)List of 3
$ baseAgrosyst :List of 6
..$ identifiantRechercheComparables: chr [1:4] "domaine_code" "sdc_code" "culture_destination" "domaine_campagne"
..$ quantitative : chr [1:21] "zone_surface" "sum_irrigation_zone_cor" "IRRI_dose_moy" "N_miner_Year" ...
..$ qualitative :List of 19
.. ..$ domaine_campagne : chr [1:25] "2001" "2002" "2003" "2004" ...
.. ..$ culture_agregee : chr [1:255] "Ail" "Artichaut" "Asperge" "Asperge / Courgette" ...
.. ..$ destination_simple_cor : chr [1:10] "Autre" "Cosse" "Ensilage" "Fibre" ...
.. ..$ sdc_type_agriculture_simple: chr [1:2] "BIO" "CONVENTIONNEL"
.. ..$ INSEE_COM : chr [1:1988] "01007" "01025" "01027" "01049" ...
.. ..$ codeDepartement : chr [1:88] "01" "02" "03" "04" ...
.. ..$ otex_18_nom : chr [1:16] "" "Autres associations" "Autres herbivores" "Bovins lait" ...
.. ..$ sdc_filiere : chr [1:2] "GRANDES_CULTURES" "POLYCULTURE_ELEVAGE"
.. ..$ type_agriculture : chr [1:4] "Agriculture biologique" "Agriculture conventionnelle" "En conversion vers l'agriculture biologique" "Information obligatoire"
.. ..$ culture_type : chr [1:2] "DEROBE" "PRINCIPALE"
.. ..$ Tag_CROPS_MixOfVariety : chr [1:2] "non" "oui"
.. ..$ unite_cor : chr [1:7] "HL_HA" "Q_HA" "Q_HA_TO_STANDARD_HUMIDITY" "TONNE_HA" ...
.. ..$ Campagne_INSEE_COM : chr [1:11445] "2001_21106" "2002_21106" "2003_21106" "2004_21106" ...
.. ..$ grand.région : chr [1:11] "" "BP - ile de France" "bretagne" "centre-est" ...
.. ..$ codeGrandeRegion : chr [1:11] "1" "10" "11" "2" ...
.. ..$ precedent_agregee : chr [1:389] "Ail" "Arbres et arbustes d'ornement / Phacélie / Plantes à parfum / Radis / Tournesol / Trèfle d'alexandrie / Vesce porte-graine" "Asperge" "Association céréale-Légumineuse" ...
.. ..$ culture_destination : chr [1:1979] "" "Ail" "Ail (Autre)" "Ail (Grain)" ...
.. ..$ irrigation : chr [1:2] "non" "oui"
.. ..$ dataBase : chr "agrosytDataBase"
..$ binaire :List of 2
.. ..$ Tag_CROPS_MixOfVariety:List of 2
.. ..$ irrigation :List of 2
..$ date : chr "date_recolte"
..$ allCol : chr [1:46] "domaine_code" "sdc_code" "domaine_campagne" "culture_agregee" ...
$ baseDetailSubstanceActive:List of 2
..$ quantitative: chr "dose_sa_G_HA"
..$ qualitative : chr "nom_sa"
$ baseDetailIntrant :List of 1
..$ quantitative: chr [1:3] "INSECTICIDAL" "HERBICIDAL" "FUNGICIDAL"3.3 Les paramètres
La description des différents paramètres est disponible dans le Swagger de l’API MoCoRiBA.
Étant donnée la diversité des possibilités de comparaisons pour trouver des exploitations comparables, il y a beaucoup de paramètres, même si plusieurs sont peu utilisés.
3.3.1 Descriptions approfondie de la construction de quelques paramètres
- niveauSimilarite :
Niveau de calcul de similarité et de recherche des exploitations comparables.
Pour définir le niveau de calcul, incluez dans un vecteur les éléments choisis parmi la liste suivante : - “domaine_code” : Travail sur des exploitations individuelles (toujours présent par défaut). - “sdc_code” : Travail par système de culture (à utiliser si plusieurs systèmes sont définis au sein d’une même exploitation). - “culture_destination” : Travail au niveau culture et destination (si absent, au niveau du système de culture). - “precedent_agregee” : Si le travail se fait à l’échelle de la culture, intègre le précédent cultural. - “domaine_campagne” : Travail par campagne de culture (si absent, la période climatique est définie par le paramètre PlageClimatique).
Exemples :
niveauSimilarite <- c(‘domaine_code’, ‘culture_destination’, ‘domaine_campagne’)
=> on recherche des exploitations comparables par culture de l’exploitation et
par campagne de culture
niveauSimilarite <- c(‘domaine_code’, ‘domaine_campagne’)
=> on recherche des exploitations comparables par campagne de culture
niveauSimilarite <- c(‘domaine_code’)
plageClimatique <- c(2015,2025)
=> on recherche des exploitations comparables sur la période 2015-2025
niveauSimilarite <- c(‘domaine_code’,’culture_destination’)
plageClimatique <- c(2015,2025)
=> on recherche des exploitations comparables sur la période 2015-2025
(quelque soit les campagnes de cultures sur mon exploitation)
- niveauSortie :
La construction est identique au paramètre niveauSimilarite ci-dessus et dans le cas général on utilise le même vecteur.
- typeComparaison :
Critère de similarité permettant de définir la méthode utilisée pour sélectionner les exploitations comparables :
- “faibleEcartCond” : méthode de la distance environnementale (voir la documentation Viz: La distance environnementale).
- “region_agroclimatique” : même grande région (voir la documentation Viz : Les grandes régions agroclimatique).
- “National” : ensemble du territoire national.
- “communes” : liste de commune spécifique (accessible uniquement en interne).
- preSelBaseComp (filtres additionels) :
Permet de garder ou d’exclure des exploitations/parcelles … avec des filtres superposés et plus ou moins complexes sur les variables qualitative.
Cette variable est une liste de listes de listes dont les noms correspondent à des colonnes qualitatives de la table de données tableAnnuelle (voir /StructureBaseAgrosyst).
Si les deux listes emboitées ont le même nom, cela correspond à une sélection simple de toutes les parcelles qui correspondent au filtre décrit dans la deuxième liste : - type : inclure ‘include’ ou exclure ‘exclude’ la sélection
- values : valeurs sélectionnées dans la colonne (voir /StructureBaseAgrosyst)
- all : pour les différentes lignes ayant même valeurs dans le nom de la liste de premier niveau garde/exclue celles qui ont l’une ou l’autre des éléments dans values (TRUE) ou tous les éléments dans values (FALSE)
L’exemple ci-dessous illustre plusieurs typologies de construction possibles. Il est possible de cumuler les filtres pour deux premiers niveaux de la liste.
preSelBaseComp <- list(
'filiere' = list('filiere'=list('filiere'=list(type = 'include',all=FALSE,values = c('GC', 'PC'))), # On conserve les filieres GC 'OU' PC
'region' = list('region'=list('region'=list(type = 'exclude',all=TRUE,values = c('North', 'East'))), # On conserve les regions autres que North et East
'domaine_code' = list('culture'=list(type='include',all=FALSE, values=c('BTH','Colza'))), # On conserve les exploitations avec du BTH 'OU' du Colza
'domaine_code' = list('culture'=list(type='include',all=TRUE, values=c('BTH','Colza'))), # On conserve les exploitations avec du BTH 'ET' du Colza
'domaine_code' = list('culture'=list(type='exclude',all=FALSE, values=c('BTH','Colza'))) # On conserve les exploitations qui n'ont pas de BTH 'OU' de Colza
'domaine_code' = list('culture'=list(type='include',all=TRUE, values=c('BTH','Colza')),
'filiere'=list(type='include',all=TRUE,values=c('BIO'))) # On conserve les exploitations avec du BTH 'OU' du Colza et qui ont une filière BIO
)
- selUserQuali (filtres additionels) :
Identique à la construction de preSelBaseComp si dessus.
- selUser
Permet de filtrer les variables quantitatives à l’échelle de la sortie. Ce filtre s’applique aux sorties de l’exploitation de référence et des comparables. Par exemple, cela signifie que pour une sortie à l’échelle d’une culture (qui agrège donc plusieurs parcelles), le filtre s’applique sur le résultat de l’agrégation des données des parcelles.
Cette variable est une liste dont les noms correspondent à des colonnes quantitatives de la table de données tableAnnuelle (voir /StructureBaseAgrosyst)
Exemple :
selUser <- list("IFT_total"=c(0,3.5),
"FET_N_total"=c(50,120))3.3.2 Paramètrage pour l’exemple
Ci-dessous, nous listons l’ensemble des paramètres utilisés pour l’exemple des appels de fonctions dans la suite de ce document.
Paramètres pouvant permettre de définir l’exploitation :
dfRef <- datExpFictive # Exploitation 'fictive' créer précédement
INSEE_COM <- "78615" # Code insee de la commune de l'exploitation
campagnes <- c(2020:2023) # campagne de culture de l'exploitation
culture_destination <- c("Blé tendre d'hiver (Grain)","Tournesol (Grain)","Maïs (Grain)") # Cultures de l'exploitationParamètres pour spécifier la méthode de recherche des comparables :
niveauSimilarite <- c("domaine_code","sdc_code","culture_destination","domaine_campagne") # On va rechercher des exploitation comparaible au niveau de culture pour des camapgnes spécifiques
plageClimatique <- NULL # Paramètre utilisé lorsque l'on recherche des comparables à l'échelle du climat et non par campagne
typeComparaison <- "faibleEcartCond" # On utilise la méthode de la distance environnementale
distBasedDistSlider <- "nParc"
seuilDist <- 5 # si distBasedDistSlider="nParc", pourcentage de parcelles les plus similaires
# si distBasedDistSlider="distance", distance environnementale euclidienne seuil
forceMatchYear <- FALSE # en distance environnementale, ne prends des similaires que sur la même campagne. Dans notre cas non.
# On sélectionne uniquement les exploitations en agriculture conventionelle
preSelBaseComp <- list("sdc_type_agriculture_simple"=
list("sdc_type_agriculture_simple"=
list(
"type"='include',
"values"="CONVENTIONNEL",
"all"=FALSE)))
uuid_sel <- NULL # Permet d'identifier des parcelles spécifiques et sera définit plus tard.
culture_destination_list <- NULL # Spécifier la listes des cultures et destinations d'intérêt pour l'utilisateur au format Agrosyst : "Blé tendre d'hiver (Grain)", "Colza d'hiver (Grain)", ... (voir /culturesDisponibles). Se combine avec les paramètres uuid et campagnes.
campagnes_list <- NULL # Spécifier la listes des campagnes d'intérêt pour l'exploitation de référence (Ex [2020,2022,2023]). Se combine avec les paramètres uuid et culture_destination.Paramètres pour les sorties de la comparaison :
variablesSortie <- c("rendZone","IFT_total") # Les variables de pratiques et de résultats qui nous intéressent
pressionBioagresseurs <- FALSE # On ne veut pas récupérer également les pressions de différents bioagresseurs des cultures
niveauSortie <- niveauSimilarite # Le niveau de sortie sera identique au niveau de recherche des comparables
selUser <- NULL # Pas de sélection sur les variables quantitatives à postériorie sur le résultats des comparables
selUserQuali <- NULL # Pas de sélection sur les variables qualitative à postériorie sur le résultats des comparables
variablesSortieDetailsIntrants <- c("INSECTICIDAL","nom_sa","dose_sa_G_HA") # Variables qui nous intéressent pour plus de détail
time_scale <- "mois" # Echelle du détail au niveau mensuelleParamètre d’état de la session utilisateur :
typeExpire <- "temporaire"3.4 Effectuer une comparaison avec DEPHY
Il existe deux méthodes pour réaliser une analyse menant au même résultat, mais adaptées à des besoins différents. Ces méthodes seront détaillées ci-dessous sous la forme de deux cas d’usage de l’API :
- Cas 1 : Je souhaite effectuer un nombre restreint d’analyses sur une même exploitation.
- Cas 2 : Je souhaite effectuer un nombre plus important d’analyses sur une même exploitation.
L’ensemble des paramètres disponibles pour chaque fonction vous permet d’ajuster votre analyse en fonction de vos critères. Pour illustrer ces démarches d’analyse, nous utiliserons une exploitation fictive et le paramétrages définie dans la partie précédente.
3.4.1 Cas 1 : Je souhaite effectuer un nombre restreint d’analyses sur une même exploitation
Dans le cas où vous souhaitez effectuer un nombre très restreint d’analyses sur une même exploitation, la solution la plus simple consiste à faire un appel direct à l’API. Pour cela, il suffit de fournir l’ensemble des informations et paramètres nécessaires :
- les informations sur l’exploitation de référence,
- les paramètres pour la recherche des exploitations comparables,
- les paramètres de sortie de l’analyse.
Cela vous permettra de récupérer directement les résultats de l’analyse.
Si vous disposez des données d’une exploitation réelle, utilisez la fonction /GetComparaisonExploitation. Si vous ne disposez pas de données d’une exploitation réelle, vous pouvez directement utiliser la fonction /GetComparaisonCodeInsee. Celle-ci intègre la fonction /basicAgrosystLine pour créer une exploitation dite ‘fictive’.
À Noter : L’utilisation du résultat de /basicAgrosystLine directement dans /GetComparaisonExploitation produira le même résultat.
Exemple d’utilisation de la fonction /GetComparaisonCodeInsee :
body <- list(INSEE_COM = INSEE_COM,
campagnes = campagnes,
cultures = culture_destination,
plageClimatique = plageClimatique,
niveauSimilarite = niveauSimilarite,
typeComparaison = typeComparaison,
seuilDist = seuilDist,
forceMatchYear = forceMatchYear,
distBasedDistSlider = distBasedDistSlider,
preSelBaseComp = preSelBaseComp,
selUser = selUser,
selUserQuali = selUserQuali,
pressionBioagresseurs = pressionBioagresseurs,
variablesSortie = variablesSortie,
niveauSortie = niveauSortie
)
resultAnalyseExpFict <- BasicPostMoCoRiBA(functionName="GetComparaisonCodeINSEE",
body=body,
token=tokenFunctionsAPI
)Voir le résultat et son analyse dans la partie Analyse de la sortie
Exemple d’utilisation de la fonction /GetComparaisonExploitation à partir d’une exploitation fictive :
body <- list(dfRef=dfRef,
plageClimatique = plageClimatique,
niveauSimilarite = niveauSimilarite,
typeComparaison = typeComparaison,
seuilDist = seuilDist,
forceMatchYear = forceMatchYear,
distBasedDistSlider = distBasedDistSlider,
preSelBaseComp = preSelBaseComp,
selUser = selUser,
selUserQuali = selUserQuali,
pressionBioagresseurs = pressionBioagresseurs,
variablesSortie = variablesSortie,
niveauSortie = niveauSortie
)
resultAnalyseExpFict<- BasicPostMoCoRiBA(functionName="GetComparaisonExploitation",
body=body,
token=tokenFunctionsAPI
)Voir le résultat et son analyse dans la partie Analyse de la sortie
3.4.2 Cas 2 : Je souhaite effectuer un nombre plus important d’analyses sur une même exploitation
Si vous souhaitez travailler sur une même exploitation en effectuant plusieurs comparaisons, ou si vous prévoyez de reprendre votre analyse ultérieurement dans un délai relativement court, vous pouvez utiliser le système de session utilisateur. Ce système offre plusieurs avantages :
- Réduction des envois de données répétées (notamment celles de l’exploitation de référence).
- Éviter la répétition de certaines opérations de calcul (dans le cas d’appelles séparés pour différente sortie mais basé sur le même paramétrage de recherche de comparables).
- Possibilité de reprendre plus tard l’analyse de votre exploitation.
a - Demander l’ouverture d’une session utilisateur pour récupérer un id_session et password avec /OpenUserSession
userSession <- BasicGetMoCoRiBA(functionName = "OpenUserSession",
token = tokenFunctionsAPI) # Autorise l'accès à la fonction
id_session <- userSession[["id_session"]]
passwordSession <- userSession[["password"]]
print(userSession)$id_session
[1] "mVQxxhNdpubjJmYlPovE"
$password
[1] "w68TRTckm9B0ilWoNbEz"b - Pour transmettre les données de l’exploitation de référence à l’API, utilisez la fonction /SaveFarmSession (que ces données concernent une exploitation réelle ou fictive).
Cette fonction permet à l’API :
- D’ajouter éventuellement certaines informations manquantes aux données transmises.
- De générer un identifiant unique (uuid) pour chaque ligne du dataframe (par exemple, équivalent à un identifiant spécifique à chaque parcelle).
Utilité de cet identifiant : Il pourra être utilisé par la suite pour préciser sur quelles données (parcelles) vous souhaitez travailler.
body <- list(id_session = id_session,
dfExploitations = dfRef,
tableDetailIntrants = NULL, # Facultatif
tableDetailPhytoSubstancesActives = NULL # Facultatif
)
farmSave <- BasicPostMoCoRiBA(functionName = "SaveFarmSession",
body = body,
token = tokenFunctionsAPI)
str(farmSave,1)List of 1
$ tableAnnuelle:'data.frame': 2 obs. of 21 variables:Pour la suite, il est possible de limiter l’analyse à un sous-ensemble de parcelles, en sélectionnant certaines parcelles spécifiques, une ou plusieurs cultures et/ou une ou plusieurs campagnes.
Dans cet exemple, nous choisissons de travailler sur 2 cultures et 2 campagnes spécifiques. Nous récupérons ensuite les identifiants (uuid) des parcelles correspondantes pour cibler notre analyse.
uuid_sel <- farmSave$uuid[which(farmSave$culture_destination %in% c("Blé tendre d'hiver","Maïs (Grain)") &
farmSave$domaine_campagne %in% c(2022,2023))]c - Deux fonctions séparées permettent de gérer la recherche de comparables et les sorties des analyses.
c1 - Effectuer une recherche de comparable avec la fonction /TriageExploitation.
La fonction retournera ‘TRUE’ une fois la recherche d’exploitation comparables réalisées.
body <- list(id_session = id_session,
uuid_list = uuid_sel,
culture_destination = culture_destination_list,
campagnes = campagnes_list,
niveauSimilarite = niveauSimilarite,
typeComparaison = typeComparaison,
distBasedDistSlider = distBasedDistSlider,
seuilDist = seuilDist,
forceMatchYear = forceMatchYear,
plageClimatique = plageClimatique,
selBaseCompQuali = preSelBaseComp
)
triageReady <- BasicPostMoCoRiBA(functionName = "TriageExploitation",
body = body,
token = tokenFunctionsAPI)
print(triageReady)$ready
[1] TRUE
$id_etat_session_temp
[1] "Byt4tvgJTZ"L’état de la session, défini par le paramétrage actuel de la recherche des exploitations comparables, est identifié par un id_etat_session_temp. Ce paramètre permet de garantir que, lors des appels aux fonctions suivantes, la référence au bon paramétrage de recherche des comparables soit maintenue.
Note : Il n’est actuellement pas possible de récuperer un état de session antérieur.
c2 - Pour récupérer les résultats de l’analyse, utilisez les fonctions /GetAnalyseAnnuelle et /GetDetailsIntrants
Ces fonctions peuvent être appelées plusieurs fois afin d’obtenir différentes sorties, tout en se basant sur une même recherche d’exploitations comparables.
La fonction /GetAnalyseAnnuelle permet d’effectuer une comparaison annuelle des résultats et des pratiques des exploitations.
body <- list(id_session = id_session,
selUser = selUser,
QualiSelUser = selUserQuali,
pressionBioagresseurs = pressionBioagresseurs,
variablesSortie = variablesSortie,
niveauSortie = niveauSortie
)
analyseParc <- BasicPostMoCoRiBA(functionName = "GetAnalyseAnnuelle",
body = body,
token = tokenFunctionsAPI)Voir le résultat et son analyse dans la partie Analyse de la sortie
La fonction /GetDetailsIntrants permet une analyse plus détaillée à différentes échelles temporelles, notamment au niveau mensuel. Elle permet aussi travailler à la substance active pour les produits phyto-phamaceutiques.
body <- list(id_session = id_session,
variablesSortie = variablesSortieDetailsIntrants,
niveauSortie = niveauSortie,
time_scale = time_scale
)
analyseDetails <- BasicPostMoCoRiBA(functionName = "GetDetailsIntrants",
body = body,
token = tokenFunctionsAPI)Voir le résultat et son analyse dans la partie Analyse de la sortie
d - Il est possible de faire une comparaison directement avec les sortie, sans passer par les 2 fonctions précédentes avec la fonction /ComparaisonExploitationSession
On fournit simplement les informations de session pour que le système API puisse récupérer l’exploitation de référence.
body <- list(
# Paramètres de triage :
id_session = id_session,
uuid_list = uuid_sel,
culture_destination = culture_destination_list,
campagnes = campagnes_list,
niveauSimilarite = niveauSimilarite,
typeComparaison = typeComparaison,
distBasedDistSlider = distBasedDistSlider,
seuilDist = seuilDist,
forceMatchYear = forceMatchYear,
plageClimatique = plageClimatique,
preSelBaseComp = preSelBaseComp,
# Paramètres de sorties :
niveauSortie = niveauSortie,
variablesSortie = variablesSortie,
pressionBioagresseurs = pressionBioagresseurs,
selUser = selUser,
selUserQuali = selUserQuali
)
analyseDirect <- BasicPostMoCoRiBA(functionName = "ComparaisonExploitationSession",
body = body,
token = tokenFunctionsAPI)Voir le résultat et son analyse dans la partie Analyse de la sortie
e - D’autres fonctions permettent de gérer le statut de la session et de récupérer des informations et des paramètres
e1 - La fonction /updateExpireUserSession permet de modifier la durée de vie d’une session.
Session temporaire (par défaut) : La session est automatiquement supprimée 2 heures après la dernière opération.
Session persistante : La session est supprimée après 7 jours d’inactivité (dernière opération).
sessionLife <- BasicGetMoCoRiBA(functionName = "updateExpireUserSession",
id_session = id_session,
typeExpire = typeExpire,
token = tokenFunctionsAPI)
print(sessionLife)$message
[1] "La session sera definitivement fermée dans 2 heures."e2 - La fonction /closeUserSession permet d’indiquer dans les paramètres d’état que l’utilisateur n’utilise plus la session.
Attention : Cette fonction ne supprime pas la session, elle se contente de signaler sa fermeture.
delaiAvantSuppression <- BasicGetMoCoRiBA(functionName = "CloseUserSession",
id_session = id_session,
token = tokenFunctionsAPI)
print(delaiAvantSuppression)$message
[1] "La session sera definitivement fermée dans 2 heures."e3 - La fonction /ReloadFarmSession permet de récupérer le dernier état enregistré de la session, incluant :
- Les paramètres utilisés pour la recherche d’exploitations comparables.
- Les données des exploitations.
Important : Les données de l’exploitation ne seront retournées que si le mot de passe de la session est fourni.
reloadFarm <- BasicGetMoCoRiBA(functionName = "ReloadFarmSession",
id_session = id_session,
password = passwordSession, # Facultatif
token = tokenFunctionsAPI)
str(reloadFarm,1)List of 12
$ id_session : chr "mVQxxhNdpubjJmYlPovE"
$ niveauSimilarite : chr [1:4] "domaine_code" "sdc_code" "culture_destination" "domaine_campagne"
$ typeComparaison : chr "faibleEcartCond"
$ distBasedDistSlider: chr "nParc"
$ seuilDist : int 5
$ forceMatchYear : logi FALSE
$ plageClimatique : list()
$ preSelBaseComp : list()
$ sessionExpireStatus: chr "temporaire"
$ sessionStatus : chr "close"
$ dfExploit :'data.frame': 2 obs. of 21 variables:
$ uuid_list : list()3.5 Analyse de la sortie des résultats de comparaison
Les sorties des fonctions suivantes sont construites sur la même base :
/GetComparaisonCodeInsee
/GetComparaisonExploitation
/GetAnalyseAnnuelle
/GetDetailsIntrants
/GetComparaisonExploitationSession
3.5.1 Premier niveau de la sortie
La sortie est une liste d’éléments dont le contenu peut varier selon que vous utilisiez le système de session ou non, ou en fonction du paramétrage de la similarité. Les résultats proprement dits se trouvent dans l’élément res.
List of 11
$ sessionStatus : # Elément de session : Si la session est en cours d'utilisation ("open") ou non ("close")
$ id_etat_session_temp: # Elément de session : Id correspondant au paramétrage actuel de la recherche de comparables
$ niveauSimilarite : # critère de similarité (échelle de la similarité)
$ typeComparaison : # critère de similarité
$ distBasedDistSlider : # critère de similarité
$ seuilDist : # critère de similarité
$ forceMatchYear : # critère de similarité
$ namesBasesComp : # Nom de la base comparaison
$ res : **# Résultats de la comparaison**
$ Echelons : # Structure de 'res'
$ warnings : # Eventuelle message de problèmesExemple :
str(analyseParc,1)List of 10
$ sessionStatus : chr "open"
$ id_etat_session_temp: chr "Byt4tvgJTZ"
$ niveauSimilarite : chr [1:4] "domaine_code" "sdc_code" "culture_destination" "domaine_campagne"
$ distBasedDistSlider : chr "nParc"
$ seuilDist : int 5
$ forceMatchYear : logi FALSE
$ namesBasesComp : chr "agrosyst"
$ plageClimatique : list()
$ res :List of 1
$ Echelons : chr [1:4] "domaine_code" "sdc_code" "culture_destination" "domaine_campagne"3.5.2 Structuration des résultats dans l’élément res
L’élément res qui contient les résultats de l’analyse est une liste de listes à plusieurs niveaux. Le nombre de niveau dépend du paramètre de sortie niveauSortie. En effet, si une analyse est réalisée sur plusieurs cultures et/ou campagnes (ou autres possibilités), il y aura forcément un résultat par culture et/ou par campagne. L’élément Echelons donne la correspondance de chaque niveau de la liste.
Dans l’exemple, l’arrangement se fait par exploitation (1er niveau), culture (2ème niveau) et campagne (3ème niveau).
str(analyseParc[["res"]][["exploitation_fictive_code_insee"]][[1]][["Blé tendre d'hiver (Grain)"]][["2021"]],1)$ nbSimilaires : int 427 # Nombre de comparables
$ qualifiant_similaires: chr "culture x campagne" # 'Unité' des comparables d'après l'échelle de la sortie (Voir ci-dessous)
$ distEnvRef : num 15.2 # En distance environnementale, valeur de référence
$ distEnvReal : num 9.86 # En distance environnementale, valeur max de la distance des comparables sélectionées
$ propBase : int 5 # Proportion de la base de comparaison sélectionnée
$ rendZone :List of 10 # Résultats pour les rendements
$ IFT_total :List of 9 # Résultats pour les IFT
$ ... :List of . # Résultats autres indicateurs
Selon le niveau de sortie choisi, un comparable peut être défini à différentes échelles (*qualifiant_similaires*) :<br>
"Système × Période climatique" : Un système considéré sur la période climatique définie.<br>
"Culture × Période climatique" : Une culture dans un système donné, sur la période climatique définie.<br>
"Système × Campagne" : Un système pour une campagne donnée.<br>
"Culture × Campagne" : Une culture pour une campagne donnée.<br>
"Grandes parcelles" : Une culture associée à un précédent cultural pour une campagne donnée.<br>La liste résultat de chaque indicateur a une structure identique. L’élément IFT_total se décompose par exemple en :
$ moyPondReference : num 2.17 # Moyenne pondérée par les surfaces de potentiellement plusieurs parcelles pour l'exploitation de référence
$ posQuantileReference: num 0.576 # Position quantile de "moyPondReference" dans la distribution des comparables
$ quantilesSimilaires : num [1:5] 0.539 1.143 1.736 2.557 4.175 # Valeurs des quantiles de la distribution des comparables (5%, 25%, 50%, 75%, 95%)
$ meanSimilaire : num 1.97 # Moyenne simple de potentiellement plusieurs parcelles pour l'exploitation de référence
$ BonMedian : num 1.14 # Médiane des performants dans les comparables : ceux qui sont dans les 50% inférieurs pour les IFTs totaux et 50% supérieur en rendement
$ MauvMedian : num 2.87 # Médiane des améliorables (50% supérieur en IFT, 50% inf en rendement)
$ propNAsurfacique : int 0 # Proportion de surface des similaires pour lesquelles il n'y a pas de donnéesNotez que pour les rendements, il y a un élément supplémentaire qui permet de connaître l’unité des rendements :
$ unite_rdt : chr "Q_HA_TO_STANDARD_HUMIDITY"Il peut aussi y avoir un élément suplémentaire lorsque l’on demande le détail des substances actives pour la fonction /GetDetailsIntrants :
$ substance_active : 'data.frame': 1 obs. of 3 variables:3.6 Exemple résumé sans R
3.6.1 Récupérer les données d’une exploitation de test
GET https://mocoriba.fr/mocoribaAPI/GetAgrosystTableForWiuzIdExp?idExp=**********token=**********
GET https://mocoriba.fr/mocoribaAPI/GetAgrosystFarmData?idExp=**********token=**********Qui retourne un json avec trois tables : - tableAnnuelle : synthèse des pratiques réalisées sur chaque parcelle par campagne.
- tableDetailIntrants : données de traitements phyto-pharmaceutiques par mois de l’année.
- tableDetailPhytoSubstancesActives : données de traitements phyto-pharmaceutiques à la substance active par mois de l’année.
{"tableAnnuelle":[
{
domaine_code : chr "**********", # Id de l'exploitation
domaine_nom : chr "**********", # Nom de l'exploitation
sdc_code : chr "**********", # Id du système de culture
zone_id : chr "**********", # Id de la parcelle
long : num "*****" # Longitude de la parcelles
lat : num "*****" # Latitude de la parcelles
domaine_campagne : int 2020, # année de récolte (campagne)
grande.région : chr "sud-ouest", # région agro-climatique, voir (https://mocoriba.fr/documentation/donnees_methodes_MoCoRiBAviz.html#GR_agroCLim)
codeGrandeRegion : num 11, # id région agro-climatique
codeDepartement : chr "64", # Code du département
INSEE_COM : chr "64554", # Code INSEE de la commune
zone_surface : num 5.01, # Surface de la parcelle (en hectares)
culture_agregee : chr "Maïs", # Culture d'après le référentiel Agrosyst
destination_simple_cor : chr "Grain", # Destination de la récolte d'après le référentiel Agrosyst
culture_destination : chr "Maïs (Grain)", # culture_agregee (destination_simple_cor),
culture_type : chr "PRINCIPALE" # Type culture (principale, intermédiaire, IAE, jachère,...)
precedent_agregee : chr "Féverole d'hiver", # Culture précédente au format Agrosyst
N_miner_Year : num 0, # Apports d'azote minérale en unités d'azote
N_org_Year : num 50, # Apports d'azote organique en unités d'azote
FERT_N_total : num 50, # Apports d'azote organique + minérale
TILL_Labour : num 1, # Nombre de labours
TILL_nbAvSemis : num 1, # Nombre de passages de travail du sol avant semis (sauf labour)
TILL_nbApSemis : num 0, # Nombre de passages de travail du sol après semis
TILL_total : num 2, # Nombre total de travail du sol
irrigation : chr "non", # Culture irriguée ou non
sum_irrigation_zone_cor : num NA, # Quantité d'eau apporté (en mm)
rendZone : num 110, # Rendement de la culture
unite_cor : chr "q/ha", # Unité du rendement de la culture
date_recolte : Date "2020-10-30", # Date de récolte
indice_Shannon_sdcCamp : num 0.683, # Diversité cultivée dans le système de culture pour cette campagne (indice de Shannon)
exp_indice_Shannon_sdcCamp: num 1.98, # Exponnetiel de l'indice de Shannon (homogène à un nombre de culture présente à proportion également dans le système)
IFT_INS_Year_Agrosyst : num 1, # IFT insecticides annuel (recalculé pour être homogène à Agrosyst)
IFT_HER_Year_Agrosyst : num 1.35, # IFT herbicides annuel (recalculé pour être homogène à Agrosyst)
IFT_FUN_Year_Agrosyst : num 0, # IFT fongicides annuel (recalculé pour être homogène à Agrosyst)
IFT_n_pass_total : num 2, # Nombres de passages herbicides, fongicides ou insecticides
IFT_total : num 2.35, # IFT insecticides + fongicides + herbicides
},
{idem pour autres années, cultures, parcelles}
],
"tableDetailIntrants": [
{
zone_id : chr "**********", # Id de la parcelle
zone_surface: num 0.96, # Surface de la parcelle (en hectares)
mois : int 5, # Mois (janvier=1, ...)
campagne : int 2023, # Année de récolte (campagne)
INSECTICIDAL: num 0, # IFT insecticides
HERBICIDAL : num 0, # IFT herbicides
FUNGICIDAL : num 0, # IFT fongicides
},
{idem pour autres parcelles, mois}
],
"tableDetailPhytoSubstancesActives": [
{
zone_id : chr "**********", # Id de la parcelle
mois : int 4, # Mois (janvier=1, ...)
campagne : int 2020, # Année de récolte (campagne)
intrant_phyto_type: chr "HERBICIDAL", # Type de produit phyto-pharmaceutique
nom_sa : chr "benoxacor", # Nom de la substance active utilisée
dose_sa_G_HA : num 90, # Quantité épandue par hectare (g/ha)
},
{idem pour autres parcelles, mois}
]
}Notez que la fonction /basicAgrosystLine permet de récupérer uniquement la table tableAnnuelle pour une exploitation sans donnée de pratique.
3.6.2 Récupérer les similaires à une exploitation de test
On utilise : /GetComparaisonExploitation
Étant donnée la diversité des possibilités de comparaisons pour trouver des similaires, il y a beaucoup de paramètres, même si plusieurs sont peu utilisés :
{
"dfRef"= tableAnnuelle, # Données de l'exploitation de référence
"plageClimatique" = NULL, # campagnes incluses, uniquement si similarité à l'échelle climatique
ex: c(2019, 2024) pour toutes les années de 2019 à 2024 incluses.
"niveauSimilarite" = ["domaine_code","sdc_code","culture_destination","domaine_campagne"], # grain de la comparaison, voir /TriageExploitation
"typeComparaison" = "faibleEcartCond", # utilise la distance environnementale pour la similarité
"forceMatchYear = "FALSE", # En distance environnementale, force à utiliser des parcelles de la même année ?
"distBasedDistSlider = "nParc", # à quoi correspond seuilDist
"seuilDist" = 5, # 5% les plus similaires
"preSelBaseComp" = {"sdc_type_agriculture_simple":
{"sdc_type_agriculture_simple":
{"type"='include',
"values"="CONVENTIONNEL",
"all"=TRUE
}
}
}, # voir ci-dessous
"variablesSortie" = ["rendZone","IFT_total","FERT_N_total","TILL_total","exp_indice_Shannon_sdcCamp"], # Variable de sortie
"pressionBioagresseurs" = FALSE, # Si l'on souhaite les variables pressions de bioagresseurs en sortie
"niveauSortie" = ["domaine_code","sdc_code","culture_destination","domaine_campagne"], # garder égal à niveauSimilarite dans le cas général.
"selUser" = NULL, # pas de sélection à posteriori sur les variables quantitatives
"selUserQuali" = NULL # pas de sélection à posteriori sur les variables qualitatives
}Pour en savoir plus sur les paramètres des fonctions, voir la partie sur Les paramètres.
3.6.3 Utilisation des sorties
Voir la partie Analyse de la sortie des résultat de comparaison.
4 Estimation de la pression ambiante des bioagresseurs
Nous avons développer des modèles de bioagresseurs pour estimer la pression ambiante des maladies et ravageurs des grandes cultures. Pour plus d’information sur la construction de ces modèles aller voir la documentation “Données et méthodes - Projet MoCoRiBA-GC” sur la partie Modélisation de la pression ambiante des bioagresseurs.
Actuellement des estimations sont disponibles pour la France métropolitaine pour 12 cultures et plus de 80 bioagresseurs.
La fonction /culturesDispoBA permet de recupérer le nom des cultures pour lesquelles des données sont disponibles.
BasicGetMoCoRiBA(functionName = "culturesDispoBA",
token = tokenFunctionsAPI)$cultures
[1] "Betterave" "Blé dur d'hiver" "Blé tendre d'hiver"
[4] "Colza d'hiver" "Maïs" "Orge d'hiver"
[7] "Orge de printemps" "Pois d'hiver" "Pois de printemps"
[10] "Pomme de terre" "Tournesol" "Triticale" La fonction /GetDefsMetriques permet de récupérer le nom des différentes métriques de maladies et ravageurs pour une cultures ainsi que leur definition.
BasicGetMoCoRiBA(functionName = "GetDefsMetriques",
Culture = "Blé tendre d'hiver",
Code_obs_gen = NULL,
token = tokenFunctionsAPI)$bioagresseurs
Code_obs_gen Organisme
1 FUSA_TIG_% Fusariose
2 HELMIN_F3 Helminthosporiose
3 OIDF3 Oïdium des céréales
4 PUC_AUT_PLANT_% Puceron vecteurs de viroses
5 PUC_EPI_PLANT_% Puceron
6 PV_% Piétin verse
7 RBF3 Rouille brune du blé
8 ROUIL_JAU_F3 Rouille jaune des céréales
9 SEPF3 Septoriose des céréales tritici
Vigicultures_protocole Type
1 Fusariose base tige_% Maladie
2 Helminthosporiose F3 Maladie
3 Oidium F3 Maladie
4 Pucerons d automne (sur plantes)_% Ravageur
5 Pucerons des épis_% Ravageur
6 Piétin verse_% Maladie
7 Rouille Brune F3 Maladie
8 Rouille Jaune F3 Maladie
9 Septoriose F3 MaladieLa fonction /BAPressionMultiCampagneINSEE_COM permet de récupérer l’estimation annuelle de la pression ambiante des bioagresseurs. Vous pouvez paramétrer la culture, les métriques souhaitées, ainsi que le format de sortie. Il est également possible d’inclure les quantiles de l’intervalle de confiance à 90 %.
À noter : Si l’information n’est pas disponible pour une année ou une code commune, l’API ne renvoie pas la ligne.
BasicPostMoCoRiBA(functionName = "BAPressionMultiCampagneINSEE_COM",
body=list(Campagne_INSEE_COM=c("2020_78615","2021_78615"),
Code_obs_gen = c("SEPF3","OIDF3")),
Culture = "Blé tendre d'hiver",
typeMatriceOutput=FALSE,
IntervalConf=TRUE,
token = tokenFunctionsAPI)$pressionBioagresseurs
INSEE_COM moy_risq Culture ID_culture_metrique Campagne
1 78615 0.1408 Blé tendre d'hiver Blé_tendre_d_hiver_OIDF3 2020
2 78615 0.0214 Blé tendre d'hiver Blé_tendre_d_hiver_OIDF3 2021
3 78615 0.2836 Blé tendre d'hiver Blé_tendre_d_hiver_SEPF3 2020
4 78615 0.1653 Blé tendre d'hiver Blé_tendre_d_hiver_SEPF3 2021
quantileQ05 quantileQ95 Code_obs_gen
1 3.4800e-02 0.2938 OIDF3
2 4.1641e-07 0.1036 OIDF3
3 1.5530e-01 0.4297 SEPF3
4 6.0700e-02 0.3022 SEPF3La fonction /BAPressionMultiCampagneMoisINSEE_COM permet de récupérer l’estimation mensuelle de la pression ambiante des bioagresseurs. Vous pouvez paramétrer la culture, les métriques souhaitées, ainsi que le format de sortie. Il est également possible d’inclure les quantiles de l’intervalle de confiance à 90 %.
BasicPostMoCoRiBA(functionName = "BAPressionMultiCampagneMoisINSEE_COM",
body=list(Campagne_INSEE_COM=c("2020_78615","2021_78615"),
Culture = "Blé tendre d'hiver",
Code_obs_gen = c("SEPF3")),
token = tokenFunctionsAPI)$pressionBioagresseursMensuelle
INSEE_COM Code_obs_gen Campagne Annee Mois moy_risq Culture
1 78615 SEPF3 2020 2020 3 0.2767 Blé tendre d'hiver
2 78615 SEPF3 2020 2020 4 0.3882 Blé tendre d'hiver
3 78615 SEPF3 2020 2020 5 0.1984 Blé tendre d'hiver
4 78615 SEPF3 2020 2020 6 0.5203 Blé tendre d'hiver
5 78615 SEPF3 2021 2021 3 0.3185 Blé tendre d'hiver
6 78615 SEPF3 2021 2021 4 0.2671 Blé tendre d'hiver
7 78615 SEPF3 2021 2021 5 0.1045 Blé tendre d'hiver
8 78615 SEPF3 2021 2021 6 0.3217 Blé tendre d'hiverDescription des informations de sortie :
{
INSEE_COM : chr "78615", # Code INSEE de la commune
Culture : chr "Blé tendre d'hiver" # Culture
Code_obs_gen : num "SEPF3", # Code de la métrique du bioagresseurs
ID_culture_metrique : chr "Blé_tendre_d_hiver_SEPF3" # Id composé de la culture et code_obs_gen
Campagne : chr "2020" # Année de récolte (campagne)
Annee : int 2020 # Année de l'observation
Mois : int 3, # Mois (janvier=1, ...)
moy_risq : num 0.2836, # Estimation de la pression
quantileQ05 : num 0.1553, # Quantile 5% de l'intervalle de confiance à 90%
quantileQ95 : num 0.4297, # Quantile 95% de l'intervalle de confiance à 90%
},
{idem pour autres communes, culture, campagne, ...}